MODÉLISATION MATHÉMATIQUE D’UN MÉCANISME INFLAMMATOIRE DANS LA SCLÉROSE EN PLAQUES
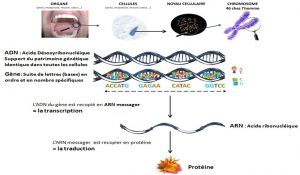
Tous les mécanismes biologiques résultent d’une interaction entre les gènes, les ARN messager, les protéines dans la cellule mais aussi de l’interaction entre les différents types de cellules de l’organisme.
L’état d’activation d’une cellule immunitaire, par exemple, dépend du taux d’expression des ARN messagers, des protéines dans la cellule qui résulte de son interaction avec son environnement. Toutes ces molécules interagissent entre elles, certaines activant, inhibant ou transformant les autres. On parle alors de réseau biologique.
Les réseaux biologiques sont des représentations de l’information globale qui circule au sein d’une cellule. Cette information est générée par des interactions physiques entre molécules mais également par une signalisation à distance grâce à des molécules sécrétées dans et à l’extérieur des cellules.
Dans une stratégie thérapeutique, il est essentiel d’identifier les acteurs « clés » d’un réseau biologique qui à eux seuls influencent l’activité globale du réseau et donc l’état d’activation de la cellule. On sait qu’il existe dans les réseaux biologiques des molécules ou des gènes dont la présence ou l’absence d’expression perturbe voire stoppe la circulation d’information au sein de la cellule conduisant à un dysfonctionnement global du réseau et du mécanisme biologique associé.
Il est donc essentiel aujourd’hui d’identifier le contexte inflammatoire qui mène à une inflammation chronique et une perte des neurones dans la sclérose en plaques. Les macrophages étant des acteurs majeurs de l’inflammation, les réseaux biologiques régulant leur état pro-inflammatoire et leur contrôle constitue une piste essentielle pour le développement de nouvelles thérapies.
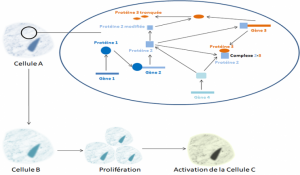
Une collaboration entre Violetta ZUJOVIC, chercheuse INSERM, neurobiologiste (Equipe « Plasticité et régénération de la myéline ») et Fabrizio DE VICO FALLANI, chercheur INRIA, biomathématicien (Equipe « ARAMIS – Algorithmes, modèles et méthodes pour les images et les signaux du cerveau humain ») vient d’aboutir à l’identification des gènes « clés » qui contrôlent l’état « pro-inflammatoire » des macrophages dans la sclérose en plaques ». Ces travaux sont publiés dans la revue scientifique Network Neuroscience.
Grâce à une stratégie de modélisation innovante appliquée sur des données d’expression d’ARN messager, les chercheurs ont pu identifier deux gènes « clés » conférant le phénotype pro-inflammatoire aux macrophages de patients, les gènes SOCS1 et SOCS3. Les gènes de la famille SOCS (suppressor of cytokine signaling) codent pour des protéines dont le rôle est d’inhiber ou d’activer les signaux induits par les cytokines, protéines centrales de l’inflammation.
D’un point de vue purement modélisation mathématique, les auteurs ont utilisé une nouvelle stratégie basée sur l’évaluation de la capacité d’un élément du réseau à contrôler un panel déterminé de cibles en affectant un rang à chaque cible. 13 cibles ont été choisies parmi les protéines sécrétées par les macrophages. Le classement des cibles (le rang) a été déterminé en fonction de la différence d’expression des ARNmessager codant pour ces protéines entre les macrophages de patients et ceux de contrôles sains.
En conclusion, ces travaux ouvrent des pistes de recherche prometteuses pour l’identification de nouvelles cibles thérapeutiques dans la sclérose en plaques ainsi que pour des stratégies de modélisation plus adaptées à l’étude des réseaux biologiques complexes sous-tendant des processus pathologiques.
https://direct.mit.edu/netn/article/5/2/337/97542/Stepwise-target-controllability-identifies
[ source ]